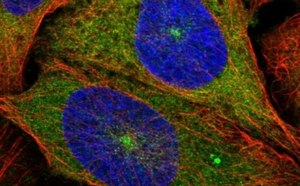
Analisi strutturale delle proteine

La funzione di una proteina è determinata direttamente dalla sua struttura, dalle sue interazioni con altre proteine e dalla sua posizione all’interno di cellule, tessuti e organi. La struttura e la funzione delle proteine, in proteomica, vengono studiate su larga scala, il che consente l’identificazione dei biomarcatori proteici associati a specifici stati patologici e rivela potenziali bersagli per le cure. La delucidazione della struttura di una proteina e la mappatura della sua localizzazione, dei relativi livelli di espressione e delle sue interazioni forniscono importanti informazioni utilizzabili per dedurne la funzione.
• Struttura proteica
• Determinazione della struttura proteica
• Mappatura proteica
Categorie in evidenza
Gli anticorpi Prestige Antibodies®, sviluppati con l'ausilio dei dati di Human Protein Atlas e convalidati con diversi metodi, costituiscono degli strumenti di ricerca affidabili.
Il nostro ampio assortimento di tamponi biologici di elevata purezza, disponibili in varie formulazioni e diversi formati, garantisce eccellente stabilità delle soluzioni e ottimo controllo del pH in tutte le fasi dei bioprocessi.
Gli isotopi stabili ISOTEC® trovano impiego negli studi di proteomica e metabolomica con traccianti, come agenti in tecniche di MRI/ MRS e in un’ampia gamma di altre applicazioni biomediche.
Disponiamo di una vasta gamma di articoli per la conservazione criogenica, tra cui rack per congelatori, fiale e provette, etichette, pennarelli e pinze, utilizzabili per la ricerca e in ambito clinico, prodotti dalle aziende più affidabili del settore.
Struttura proteica
La struttura di una proteina è determinata dalla sequenza degli amminoacidi che la costituiscono e da come la proteina si ripiega in forme più complesse.
- La struttura primaria è definita dalla sequenza di amminoacidi della proteina.
- La struttura secondaria è determinata dalle interazioni locali di segmenti della catena polipeptidica, che, via legami a idrogeno, danno luogo ad α-eliche e β-foglietti.
- La struttura terziaria definisce la conformazione tridimensionale complessiva della proteina.
- La struttura quaternaria descrive in che modo più sottounità proteiche interagiscono per formare complessi più grandi.
Determinazione della struttura delle proteine
La determinazione della struttura tridimensionale delle proteine a livello di risoluzione atomica è utile per comprenderne la funzione, per l’ingegnerizzazione di farmaci basati sulla struttura molecolare del bersaglio e per gli studi di docking molecolare.
- NMR: la spettroscopia NMR (Risonanza Magnetica Nucleare) è usata per ricavare informazioni sulla struttura e la dinamica delle proteine. Nell’NMR la disposizione spaziale degli atomi si deduce dai loro spostamenti chimici. In questa tecnica, le proteine vengono in genere marcate con isotopi stabili (15N, 13C, 2H) al fine di aumentare la sensibilità del metodo e facilitare la deconvoluzione delle strutture. I marcatori isotopici solitamente vengono introdotti nel terreno di coltura sotto forma di nutrienti marcati isotopicamente durante l’espressione proteica.
- Cristallografia ai raggi X: la cristallografia ai raggi X viene impiegata per ricavare la struttura tridimensionale delle proteine attraverso la diffrazione ai raggi X di proteine cristallizzate. I cristalli vengono fatti crescere introducendo in soluzioni proteiche altamente concentrate germi di cristallizzazione che promuovono la precipitazione e, in condizioni opportune, la formazione di cristalli ordinati di proteine. I raggi X vengono diretti sui cristalli proteici e da questi diffratti su sensori elettronici o su pellicola. Facendo ruotare i cristalli, si può registrare la diffrazione in tre dimensioni e consentire il calcolo, mediante trasformata di Fourier, della posizione di ciascun atomo nella molecola cristallina.
Mappatura delle proteine
La mappatura della localizzazione e del livello di espressione delle proteine in specifiche cellule, tessuti e organi contribuisce allo studio funzionale del proteoma. La distribuzione, nella cellula, delle proteine è decisiva per la loro funzione; errori di localizzazione o di espressione delle proteine, infatti, possono essere la causa di vari stati patologici. Progetti di mappatura come il programma Human Protein Atlas rappresentano una risorsa proteomica per la scoperta di biomarcatori e aiutano a comprendere la patologia delle malattie. La mappatura dell’interattoma serve a definire le interazioni molecolari che avvengono a livello cellulare, è di ausilio nella comprensione della funzione delle proteine e permette di individuare bersagli utili per potenziali farmaci.
Fate una ricerca tra i numerosi documenti disponibili: schede tecniche, certificati e documentazione tecnica.
Articoli tecnici correlati
- Amino acid reference chart contains the twenty amino acids found in eukaryotes, grouped according to their side chains and charge. Discover our full product line of amino acids, including Alanine, Isoleucine, Leucine, Valine, Phenylalanine, Tryptophan, Tyrosine, Aspargine, Cysteine, Glutamine, Methionine, Serine, Threonine, Aspartic acid, Glutamic acid, Arginine, Histidine, Lysine, Glycine and Proline. Learn more today.
- The human protein atlas has an aim of mapping all human proteins within cells, tissues and organs and providing open-access information to advance understanding of human biology and disease.
- Information on Isoelectric Focusing including what it is and how it is used. In order to ensure the high performance of analysis, isoelectric point (pI) standards are needed.
- We presents an informational article concerning biomolecular NMR and the use of Isotope Labeling Methods for Protein Dynamics Studies.
- Glycosylphosphatidylinisotol (GPI) anchored proteins are membrane bound proteins found throughout the animal kingdom. GPI anchored proteins are linked at their carboxyterminus through a phosphodiester linkage of phosphoethanolamine to a trimannosyl-non-acetylated glucosamine (Man3-GlcN) core.
- Visualizza tutto (10)
Protocolli correlati
- This page covers the principles and methods of chromatofocusing, a chromatography technique that separates proteins according to differences in their isoelectric point (pI).
- Protein Structural Analysis
- This protocol describes a method for chemical cross-linking of proteins using formaldehyde. With the exception of zero-length cross-linkers, formaldehyde has the shortest cross-linking span (~2-3 Å) of any cross-linking reagent, thus making it an ideal tool for detecting specific protein-protein interactions with great confidence.
Per consultare altri articoli e protocolli
Come possiamo aiutarvi
Per qualunque domanda, non esitate a inviare una richiesta di assistenza
o a chiamare il nostro Servizio Clienti:
Email custserv@sial.com
telefono +1 (800) 244-1173
Altre risorse
- Chromatogram Search
Use the Chromatogram Search to identify unknown compounds in your sample.
- Strumenti di calcolo e app
Strumenti e risorse scientifiche online per la chimica analitica, le life science, la sintesi chimica e la scienza dei materiali.
- Customer Support Request
Assistenza clienti inclusa assistenza per ordini, prodotti, account e problemi tecnici del sito web.
- FAQ
Explore our Frequently Asked Questions for answers to commonly asked questions about our products and services.
Per continuare a leggere, autenticati o crea un account.
Non hai un Account?