Projektowanie testów PCR/qPCR/dPCR
A Technical Guide to PCR Technologies
Cały proces PCR jest podatny na czynniki, które wprowadzają zmienność. Wiele zmiennych składników jest nieuniknionych, takich jak źródło próbki lub wymóg etapu odwrotnej transkrypcji. Projekt testu jest również bardzo zmienny i może stanowić różnicę między sukcesem a porażką PCR, a także przyczynia się do odtwarzalności i czułości testu. Proces projektowania testu przebiega logicznie: Pierwszym krokiem jest określenie pożądanej lokalizacji docelowej. W niektórych przypadkach sekwencja oligos jest określona przez aplikację i nie można jej uniknąć, np. wykrywanie SNP, w innych można użyć całego genu, np. określanie liczby kopii. Po wybraniu przybliżonych miejsc badania, identyfikowane są najbardziej odpowiednie startery i określane modyfikacje. Gdy testy mają być wykonywane w multipleksie, ważne jest, aby wziąć pod uwagę potencjał interakcji wszystkich oligos w reakcji, a także względną obfitość celów. W trudnych sytuacjach, np. gdy celem jest wykrycie bardzo niskiej liczby kopii lub niewielkich różnic w stężeniu celu, zaleca się wybranie i przetestowanie kilku kombinacji starterów, a następnie połączenie z odpowiednią sondą.
Proces projektowania testu jest znacznie ułatwiony dzięki zastosowaniu odpowiedniego oprogramowania do projektowania. OligoArchitect zapewnia dwie opcje wsparcia projektowania. Pierwszą z nich jest OligoArchitect Online, narzędzie do projektowania oprogramowania z szerokim zakresem opcji. Jeśli projekt wymaga specjalistycznych możliwości, drugą opcją jest zamówienie projektu za pośrednictwem OligoArchitect Consultative, korzystając z pomocy naszych ekspertów w dziedzinie biologii molekularnej.
Wybór amplikonu
Amplikon to region sekwencji docelowej, który ma być analizowany i jest objęty starterami do przodu i do tyłu PCR. Określenie wielkości amplikonu jest częściowo zależne od metody, która ma być użyta do analizy. Podczas wizualizacji fragmentów PCR za pomocą elektroforezy żelowej, fragment PCR musi być wystarczająco duży, aby można go było skutecznie wybarwić za pomocą barwnika wiążącego DNA i zmieścić się w zakresie wybranego sztucznego markera wielkości. Podobnie, podczas rozdzielania fragmentu za pomocą aparatu do elektroforezy kapilarnej, produkt PCR będzie miał wielkość od 100 par zasad do ponad 2 kb (ostatecznie ograniczony wydajnością enzymu).
Przy użyciu qPCR do końcowego odczytu wybiera się mniejszy amplikon, aby zapewnić dokładną kwantyfikację w każdym cyklu. Idealny rozmiar amplikonu qPCR wynosi od 75 do 200 zasad, chyba że ograniczenia projektowe wykraczają poza ten zakres. W rzeczywistości wielkość fragmentu może być określona przez rozważenie kilku czynników, w tym rozważanej biologii.
Lokalizacja testu może być wstępnie określona przez cele eksperymentu: Idealnie, testy do określenia obecności lub ilości docelowego mRNA są zlokalizowane nad połączeniem ekson-ekson, aby uniknąć wykrycia zanieczyszczających sekwencji gDNA. Jednak regiony te są często silnie sfałdowane; dlatego wymagana jest pragmatyczna decyzja dotycząca preferencji testu obejmującego ekson z potencjalnie gorszą wydajnością lub testu egzonowego o wyższej jakości. Jeśli mRNA jest obfity i transkrybowany z pojedynczej kopii genu, udział sygnału z zanieczyszczenia gDNA będzie znacznie mniej znaczący niż w przypadku wykrywania mało obfitego transkryptu z genu wielokopijnego. Wykrywanie SNP wymaga lokalizacji sondy lub 3' startera nad miejscem niedopasowania.
Analiza wariantów splicingu wymaga podejścia projektowego, które jest specyficzne dla celu. W niektórych przypadkach pożądane jest jednoczesne wykrycie wszystkich wariantów splicingu, co można po prostu osiągnąć, wybierając granicę eksonu, która jest zachowana między wszystkimi wariantami. Jednak badania nad zróżnicowaną ekspresją każdego wariantu splicingu wymagają bardziej kreatywnego podejścia. W jednym z przykładowych eksperymentów celem jest zbadanie, który z alternatywnych transkryptów, pokazanych na Rysunku 6.1, ulega ekspresji. Ponieważ eksony są stosunkowo małe, amplifikacja we wszystkich eksonach daje produkt około 300 zasad, z mniejszymi produktami wynikającymi z amplifikacji wariantów splicingu. Opcje projektowe dla tego badania byłyby następujące:
- Zaprojektuj kilka testów w każdym połączeniu eksonów i sonduj każdą próbkę za pomocą każdego testu.
- Zaprojektuj starter do 3' eksonu 1 i 5' eksonu 4. Amplifikacja dowolnego transkryptu składającego się z dowolnej kombinacji eksonów spowodowałaby powstanie amplikonu o określonej długości. Definicja transkryptu zostanie określona przy użyciu qPCR, krzywej topnienia barwnika SYBR Green I po reakcji.
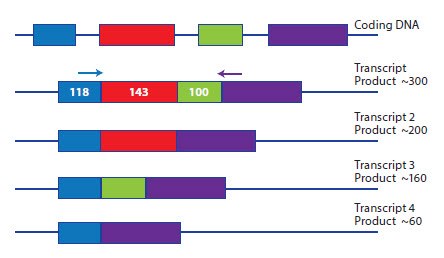
Rysunek 6.1.Schematyczne przedstawienie genu wyrażonego jako cztery potencjalne warianty splicingu. Każdy wariant splicingu można rozróżnić poprzez zaprojektowanie specyficznych par starterów w każdym połączeniu eksonów lub poprzez amplifikację z ogólnej pary starterów w eksonach 1 i 4 oraz rozróżnienie wariantów splicingu przy użyciu qPCR, analizy stopionego barwnika SYBR Green I.
Ogólnie rzecz biorąc, sekwencje amplikonów powinny być oceniane przy użyciu następujących kryteriów:
- Zaleca się wstępną ocenę regionu sekwencji docelowej.
- Upewnij się, że nie ma nieoczekiwanych SNP. Pojedyncze niedopasowanie między starterem a matrycą może obniżyć temperaturę topnienia nawet o 10 °C, wpływając na wydajność PCR.
- Potwierdź, że wybrana sekwencja nie ma homologii z żadną inną sekwencją w genomie/transkryptomie gatunku docelowego. W przypadku systemów ukierunkowanych na wiele organizmów, np, wykrywanie patogenów, określenie homologii musi obejmować wszystkie sekwencje, które mogą znajdować się w próbce.
- Sprawdź potencjał sekwencji docelowej do przyjęcia struktury drugorzędowej za pomocą algorytmu fałdowania OligoArchitect, wybranego oprogramowania do projektowania lub mfold (http://mfold.rna.albany.edu/) w celu modelowania fałdowania szablonu w żądanej temperaturze wyżarzania startera. Wybierz regiony matrycy, które mają przewidywaną otwartą strukturę, unikając struktur drugorzędowych pętli macierzystych o bardzo ujemnych wartościach ΔG. Jest to ważne przy stosowaniu jednoetapowego protokołu odwrotnej transkrypcji i starterów specyficznych dla genu.
- Unikaj sekwencji palindromicznych i regionów powtarzalnych.
- Unikaj obszarów bogatych w G/C, dążąc do około 50% zawartości GC.
- Kiedy projektowane są testy multipleksowe, amplikony powinny być jak najbardziej podobne pod względem długości i zawartości CG, aby uniknąć stronniczej amplifikacji.
- Zidentyfikuj regiony homologii lub heterogeniczności (zgodnie z wymaganiami) podczas projektowania testów dla rodzin genów. Sekwencje powinny być następnie wyrównane i zbadane pod kątem odcinków odpowiedniej sekwencji konsensusowej.
- W przypadku projektów specyficznych dla transkryptu (aby uniknąć wykrycia szablonów gDNA), w miarę możliwości należy kierować regiony docelowe na połączenie ekson-ekson.
Wybór amplikonu specyficznego dla transkryptu
Większość, ale nie całe, DNA jest eliminowane z próbki podczas oczyszczania RNA. Aby uniknąć amplifikacji DNA podczas RT-qPCR, zaleca się wybór starterów, które albo otaczają duży intron, który nie występuje w sekwencji mRNA, albo obejmują połączenie ekson-ekson (Rysunek 6.2).
Adnotacje intronów/eksonów dla znanych genów z wielu gatunków kręgowców, bakterii, protistów, grzybów, roślin i bezkręgowców metazoicznych są dostępne na stronie EnsemblGenomes (http://ensemblgenomes.org/). Alternatywnie, jeśli zarówno sekwencje genomowe, jak i cDNA dla genu docelowego są publicznie dostępne, pozycje intronów można zidentyfikować, wykonując wyszukiwanie BLAST z sekwencją cDNA względem genomowej bazy danych dla organizmu docelowego (Rysunek 6.3). Intron 1 na Rysunku 6.3 jest na tyle długi (~6,5 kb), że DNA nie powinno być amplifikowane w konwencjonalnych warunkach qPCR lub kontrolowanych warunkach PCR. Jednak wszystkie inne introny są stosunkowo krótkie (<1 kb), więc DNA prawdopodobnie będzie amplifikowane podczas RT-qPCR (na przykład Assay Optimization and Validation). Primery powinny obejmować połączenia ekson-ekson, flankować długi (kilka kb) intron lub flankować wiele małych intronów.
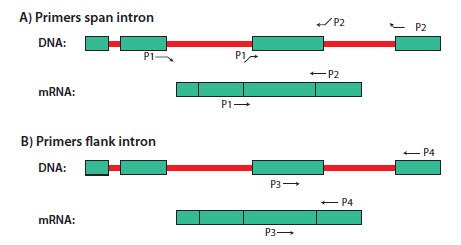
Rysunek 6.2.Ilustracja (A) starterów obejmujących introny i (B) starterów obejmujących introny do RT-PCR. Introny są zaznaczone na czerwono, a eksony na zielono. Startery P1 i P2 obejmują intron, a startery P3 i P4 otaczają intron. Należy pamiętać, że startery P1 i P2 nie wygenerują produktu PCR z DNA, chyba że temperatura wyżarzania jest bardzo niska. P3 i P4 mogą generować dłuższy produkt PCR z DNA, jeśli intron jest krótki (~ 1 kb), ale nie, jeśli jest wystarczająco długi (kilka kb).

Rysunek 6.3.Dopasowanie BLAST sekwencji cDNA do sekwencji genomowego DNA. Kompletna sekwencja cDNA dla szczurzego p53 z Genbank (numer akcesyjny NM_030989) została wykorzystana w wyszukiwaniu megaBLAST identycznych sekwencji w genomie szczura (blastn). Pokazano dopasowanie cDNA do gDNA na chromosomie 10. Korzystając z tych informacji, eksony cDNA można dopasować do odpowiednich regionów gDNA, a projektowanie starterów jest ukierunkowane na eksony, które są oddzielone długimi intronami, np. eksony 1 i 2.
Testy specyficzne dla metylacji
Metylacja DNA jest kluczową częścią różnicowania komórkowego, powodując stabilną zmianę ekspresji genów. Metylacja jest ważna dla prawidłowego rozwoju organizmów wyższych i może być dziedziczona. Regulacja genów poprzez metylację DNA polega na dodaniu grupy metylowej do pozycji 5 pierścienia pirymidynowego cytozyny lub azotu 6 pierścienia purynowego adeniny. W dorosłych tkankach somatycznych metylacja DNA zwykle występuje w kontekście dinukleotydów CpG, podczas gdy metylacja nie-CpG jest powszechna w embrionalnych komórkach macierzystych. Testy specyficzne dla metylacji wymagają identyfikacji wysp CpG w sekwencji, często w regionie promotora genu. Informacje te są automatycznie lokalizowane podczas korzystania z Beacon Designer (Premier Biosoft) i są dostępne na stronie http://www.mybioinfo.info/index.php.
Projektowanie primera
.Podczas gdy ogólne sugestie dotyczące projektowania primerów i sond opisane w tym rozdziale mają zastosowanie do wielu zastosowań, w tym badań ekspresji genów, wykrywania SNP, badań wykrywania metylacji, określania liczby kopii, monitorowania obciążenia wirusem i kwantyfikacji wariantów splicingu, każda aplikacja ma również specyficzne względy projektowe, które zostaną omówione osobno.
Ogólne kryteria projektowania starterów
Dla większości zastosowań, startery są zaprojektowane tak, aby były w pełni komplementarne do sekwencji matrycowego DNA, które mają prymować. Podstawowe rozważania projektowe dla primerów PCR obejmują:
- Prymery mają zazwyczaj długość 20-24 nukleotydów z temperaturą topnienia (Tm) około 60 °C
(59±2 °C) dla qPCR, ale mogą się różnić (55±5 °C) dla konwencjonalnej PCR. Specyficzne zastosowania mogą wymagać modyfikacji długości starterów i Tm.
- Pary starterów powinny zawierać 40-60% GC i nie powinny mieć znaczącej struktury drugorzędowej.
- Startery nie powinny być komplementarne do siebie lub starterów partnerskich, szczególnie na końcu 3'. Zmniejsza to potencjał tworzenia produktów primer-dimer podczas amplifikacji.
- Unikaj zaciskania 3' (zbadaj 5 zasad 3' i zaakceptuj 3 z nich jako A lub T i 2 jako G lub C).
- Unikaj ciągów tego samego nukleotydu, które są dłuższe niż 4 powtórzenia lub regiony palindromiczne.
Podkłady specyficzne dla SNP
.W niektórych przypadkach, takich jak projektowanie testów polimorfizmu pojedynczego nukleotydu (SNP), nie ma elastyczności co do lokalizacji testu, a otaczająca sekwencja będzie również wpływać na sekwencję wybranych oligonukleotydów. Uznanie związku między stanami klinicznymi a zarówno germinalnymi, jak i nabytymi somatycznymi SNP nadal napędza znaczne wysiłki na rzecz rozwoju coraz bardziej czułych i specyficznych systemów wykrywania. Odzwierciedla to trudny charakter dyskryminacji SNP przy użyciu hybrydyzacji oligo. Wyzwanie to wynika z różnic w destabilizacji między różnymi niedopasowaniami. Podczas gdy G:A, C:T i T:T mogą mieć silny efekt destabilizujący, G:T i C:A są znacznie słabsze, ponieważ mogą tworzyć się wiązania wodorowe, a zatem trudno jest odróżnić te pary od naturalnych G:C i T:A. Wiele systemów jest adaptacjami systemu mutacji opornych na amplifikację (ARMS)1, który był szeroko stosowany i odegrał kluczową rolę w badaniach przesiewowych w kierunku mutacji mukowiscydozy2. Startery ARMS mają długość 30 zasad (dłuższe startery, do 60 zasad są funkcjonalne). Baza na 3' jest specyficzna dla SNP, a zatem specyficzna dla sekwencji docelowej (normalna lub zmutowana baza). Dodatkowe niedopasowanie jest wprowadzane w przedostatniej pozycji. Jest to określane z uwzględnieniem sąsiednich zasad i niedopasowania SNP (Tabela 6.1 zaadaptowane z Little, 2001).
Przedrukowano za zgodą Current Protocols in Human Genetics. Little, S. 2001. Amplification-Refractory Mutation System (ARMS) Analiza mutacji punktowych. Curr. Protoc. Hum. Genet. 7:9.8.1-9.8.12.
Dodatkowe grupy badawcze wykorzystały podobne pomysły i wykazały użyteczność wprowadzania niedopasowań w pozycjach N-2 i N-3 w starterach, Liu et al.3 przeprowadzili dogłębną analizę względnych pozycji niedopasowań dla największego efektu destabilizacji, a tym samym najwyższej specyficzności.
Multiplex PCR
Amplifikacja kilku celów jednocześnie w multipleksowym PCR jest wymagana, gdy istnieje potrzeba zwiększenia przepustowości przy większej liczbie PCR na probówkę lub w celu zaoszczędzenia materiału próbki. Projektowanie starterów jest najbardziej krytycznym czynnikiem udanego multipleksowego PCR. Kluczowe jest przestrzeganie ogólnych wytycznych i sprawdzenie kompatybilności wszystkich starterów (i sond), które mają być zawarte w reakcji. W niektórych przypadkach korzystne może być użycie nieco dłuższych starterów z Tm około 65 °C. Jeśli powstałe amplikony mają być analizowane w oparciu o rozróżnianie wielkości, moc rozdzielcza analizy musi być uwzględniona w projekcie testu. Podczas próby ilościowego określenia wielu celów za pomocą qPCR, amplikony powinny być jak najbardziej podobne, aby uniknąć błędu amplifikacji. Oprócz starterów ważne jest, aby matryca nie mogła przyjąć stabilnej struktury drugorzędowej, ponieważ utrudniłoby to PCR. Jeśli wiadomo, że cele są obecne w znacząco różnych stężeniach, korzystne może być dołączenie startera blokującego do celu o wysokim stężeniu, aby ułatwić dokładne wykrycie celu o niższym stężeniu4.
Kwantyfikacja niekodującego RNA
W przeciwieństwie do genomu kodującego, szacuje się, że ~97% ludzkiego transkryptomu składa się z niekodującego RNA (ncRNA)5;6,7. Jednym z członków tej rodziny są długie niekodujące RNA, które zostały opisane jako klasa regulacyjnych cząsteczek RNA. Cząsteczki te odgrywają rolę w epigenetyce, rozwoju, nowotworach i istotnych procesach biologicznych8,9. Długie ncRNA są tradycyjnie definiowane jako składające się z nici RNA o długości co najmniej 200 zasad10,11,12. Oznacza to, że po rozpoznaniu długości amplikonu, przy projektowaniu dla tych celów nie trzeba brać pod uwagę żadnych specjalnych względów, innych niż te, o których już wspomniano.
W przeciwieństwie do tego, członkowie rodziny, którzy składają się na mikroRNA (miRNA), stanowią poważne wyzwanie projektowe. Są to krótkie niekodujące RNA (sncRNA) o długości 21-23 nt, które są wytwarzane za pośrednictwem złożonego szlaku komórkowego na kilku etapach przetwarzania transkryptu. MikroRNA negatywnie regulują translację białek poprzez wiązanie się z transkryptem (przegląd w Kato i in., 200813) i indukują tworzenie kompleksu wyciszającego indukowanego RNA (RISC)14. Komercyjne testy, takie jak MystiCq® line są pożądanym rozwiązaniem wyzwań projektowych stawianych przez miRNA. Zaproponowano kilka schematów qPCR do analizy miRNA15 a dla tych badających organizmy, dla których nie ma produktów komercyjnych, istnieje kilka publikacji opisujących potencjalne rozwiązania. Wiele z nich opiera się na dodawaniu zasad do oryginalnego miRNA poprzez ligację adaptera (rozdział 22, Casoldi, et al., w PCR Technologies; Current Innovations ed Nolan16) lub poprzez dodanie ogona poly-A przy użyciu polimerazy polyA (PAP)17. Dodanie znacznika do każdego ze starterów specyficznych dla miRNA umożliwia optymalizację czasu hybrydyzacji Tm i wykazano, że reakcje zawierające startery DNA są bardziej wydajne niż te z dodatkiem zablokowanego kwasu nukleinowego18. W tym raporcie startery DNA specyficzne dla miRNA zostały zaprojektowane przy użyciu konwencjonalnych wytycznych dotyczących starterów PCR z dodatkowymi rozważaniami:
- Zbadaj sekwencję miRNA i pomiń wszystkie końcowe zasady A w 3' - Zidentyfikuj starter do przodu jako najdłuższy odcinek sekwencji od 5' do końcowych 4 zasad 3' (ignorując zasady A zidentyfikowane powyżej).
- Preferuje się, aby jedną z 2 zasad na 3' startera forward była A lub T.
- Preferuje się, aby 3 zasady na 3' zawierały 1 lub 2 A lub T.
- Preferuje się, aby 5 zasad w 3' zawierało 2-3 A lub T.
- Zanalizuj Tm startera przedniego przy użyciu algorytmu najbliższego sąsiada. Jeśli jest poniżej 59 °C, dodaj następujące zasady do 5' w podanej kolejności i oblicz Tm po każdym dodaniu: G,A,C,G,C (otrzymując primer o postaci CGCAGN18 gdzie N to zasady specyficzne dla miRNA). Wybierz najkrótszy starter z Tm najbliższym 59 °C. Jeśli jest powyżej 59 °C, usuń zasady 5' i oblicz Tm po każdym usunięciu zasad. Wybierz najdłuższy starter z Tm najbliższym 59 °C.
- Wybierz zasady 3' dla startera odwrotnego, upewniając się, że nie są one komplementarne do startera do przodu. Oceń końcowe 5 zasad, jak opisano dla startera do przodu.
- Dodaj 15×T do startera (np, 5' T15 N5 3').
- Zanalizuj Tm startera do przodu przy użyciu algorytmu najbliższego sąsiada. Jeśli jest poniżej 59 °C, dodaj następujące zasady do 5' startera w podanej kolejności i oblicz Tm po każdym dodaniu: G,A,C,C, T,G,G,A,C, C (otrzymując primer o postaci CAGGTCCAG T15 N5 gdzie N to zasady specyficzne dla miRNA). Wybierz najkrótszy primer z Tm najbliższym 59 °C.
- Prymer RT to CAGCTCCAG T15 V N (gdzie V=A, C i G oraz N=A,C,G,T).
Przykład projektowania starterów
Ponieważ sekwencje docelowe dyktują sekwencje starterów, nie zawsze jest możliwe osiągnięcie pożądanych kryteriów projektowania. Dlatego kompromisy w projektowaniu testów są przezwyciężane przez optymalizację specyficzną dla testu. Niektóre cele PCR mogą wymagać specjalnego przetwarzania przed zaprojektowaniem udanego testu. Często spotykany przypadek dotyczy wykrywania patogenów, w tym wirusów. Powszechnie wiadomo, że wiele wirusów ma wysoki stopień zmienności w określonych miejscach w ich genomach. Dobrym przykładem jest wirus zapalenia wątroby typu B. W niedawnym badaniu, aby zaprojektować skuteczny test qPCR przeciwko znanym wariantom HBV19, konieczne było przeprowadzenie obszernego dopasowania wszystkich dostępnych sekwencji genomowych HBV. Porównano kilkaset sekwencji za pomocą ClustalW, próbując znaleźć znaczące odcinki sekwencji konsensusu, które mogłyby zostać wykorzystane do zaprojektowania ogólnego testu. Fragment wyniku wyrównania przedstawiono na Rysunku 6.4. Gwiazdki (*) reprezentują nukleotydy konsensusowe znalezione w analizie wszystkich genomów (duża liczba innych sekwencji, które były częścią tego wyrównania, nie została pokazana ze względu na ograniczenia miejsca).

Rysunek 6.4.Częściowa analiza ClustalW danych genomu HBV. Wszystkie znane sekwencje genomu HBV zostały dopasowane przy użyciu ClustalW i zidentyfikowano konserwatywne nukleotydy (*).
Przy projektowaniu starterów i sond w takich sytuacjach może być konieczne użycie oligonukleotydów, które zawierają mieszane zasady, znane również jako "wobble" lub zdegenerowane zasady. Na przykład, rozważ szczegóły sekwencji konsensusu pokazanej dla wirusa HBV (Rysunek 6.5).
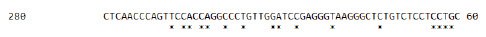
Rysunek 6.5.Wybrany region dopasowania HBV pokazujący regiony konsensusu
Region o długości około dwudziestu trzech zasad jest wymagany dla startera. W tym przypadku, biorąc pod uwagę wszystkie możliwości sekwencji dla wszystkich genomów HBV, rzeczywista sekwencja tego regionu dwudziestu trzech zasad jest pokazana na Rysunku 6.6:
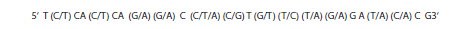
Rysunek 6.6.Permutacje sekwencji startera w celu uwzględnienia wszystkich opcji bazowych dla wybranego regionu startera konsensusowego.
Pozycje niejednoznacznych zasad można przedstawić za pomocą standardowych kodów jednoliterowych dla zasad mieszanych (Tabela 6.3). Gdy zostaną one zastosowane do sekwencji pokazanej na Rysunkach 6.5 i 6.6, oligo można opisać tak, jak na Rysunku 6.7.
A = adenozyna, C = cytydyna, G = guanozyna, T = tymidyna
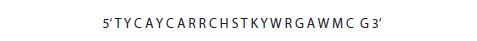
Rysunek 6.7.Niejednoznaczne zasady regionu konsensusu oligo są reprezentowane przez standardowe kody jednoliterowe.
Jest mało prawdopodobne, aby ta opcja zaowocowała udanym starterem PCR, ponieważ istnieją dodatkowe względy, które należy uwzględnić w odniesieniu do dużej liczby zdegenerowanych zasad. W szczególności, syntetyczne oligo wyprodukowane przy użyciu tej sekwencji w rzeczywistości skutkowałoby mieszaniną każdej z możliwych sekwencji pojedynczej zasady. Liczba możliwych, indywidualnych sekwencji starterów jest obliczana przez pomnożenie poszczególnych numerów zasad w każdej pozycji. Dla tej sekwencji oznacza to 1×2×1×2×1×2×1×3×2×1×2×2×1×2×2×1×2×1×2×1×1 = 6,144 możliwych indywidualnych oligos. W związku z tym efektywne stężenie każdego konkretnego oligo w reakcji jest proporcjonalnie zmniejszone. Analiza empiryczna wykazała, że liczba różnych permutacji sekwencji w starterze nie powinna przekraczać 512, dlatego ten przykład nie byłby optymalnym starterem z bazą zdegenerowaną. Przeprojektowanie w inne miejsce stanowiłoby potencjalne rozwiązanie. W przykładzie pokazanym na Rysunku 6.8, starter zawiera 2 zasady z potencjalnymi niedopasowaniami. Jednak każda z nich ma pojedynczą alternatywną zasadę, co skutkuje 4 oligonukleotydami w mieszanej syntezie, a chybotanie znajduje się w regionie 5' startera. Czynniki te zapewniają znacznie większą szansę na sukces niż starter przedstawiony na Rysunku 6.7.

Rysunek 6.8.Sekwencja pokazująca zasady konsensusu dopasowania i potencjalną lokalizację startera do regionu konsensusu. Starter konsensusowy jest pokazany za pomocą kodów wobble
Przy stosowaniu zdegenerowanych oligonukleotydów w PCR może być konieczny zmodyfikowany protokół amplifikacji. Cykle można rozpocząć od 2-5 cykli w niskiej temperaturze wyżarzania (35-45 °C). Należy również włączyć powolną rampę od temperatury wyżarzania do temperatury wydłużania, trwającą około 3-5 minut do osiągnięcia temperatury wydłużania. Protokół należy następnie zakończyć 25-40 cyklami w bardziej rygorystycznej temperaturze wyżarzania bez modyfikacji rampy.
W miarę możliwości zaleca się unikanie heterogeniczności nukleotydów. Jeśli nie jest możliwe uniknięcie regionów heterogeniczności, co często ma miejsce w przypadku trudnych celów, wówczas użycie specjalistyczne modyfikacje oligo, takie jak inozyna i inne "uniwersalne" zasady, takie jak 5-nitroindol, mogą pomóc w zmniejszeniu złożoności i dodaniu grup modyfikujących, takich jak zablokowany kwas nukleinowy (patrz Quantitative PCR and Digital PCR Detection Methods) może poprawić wydajność.
Modyfikacje starterów
Możliwe jest włączenie zmodyfikowanych nukleotydów do starterów PCR. Powszechnym przykładem jest dodanie zablokowanej zasady kwasu nukleinowego. Zablokowany kwas nukleinowy jest zmodyfikowanym nukleotydem RNA. Część rybozowa zablokowanego kwasu nukleinowego jest modyfikowana dodatkowym mostkiem łączącym tlen 2' i węgiel 4' (Ilościowa PCR i Cyfrowe metody wykrywania PCR). Mostek ten "blokuje" rybozę w konformacji 3'-endo. Zablokowany kwas nukleinowy można mieszać z zasadami DNA lub RNA w oligonukleotydzie w dowolnym miejscu. Modyfikacja Locked Nucleic Acid skutkuje zwiększoną stabilnością termiczną, umożliwiając projektowanie krótszych sond z równoważnym Tm do dłuższego, niezmodyfikowanego równoważnego startera. Sekwencje zawierające zablokowany kwas nukleinowy są bardziej specyficzne niż oligo składające się z samego DNA i idealnie nadają się do wykrywania SNP. Dodatkowe zastosowania modyfikacji Locked Nucleic Acid obejmują projektowanie oligos do analizy trudnych sekwencji, takich jak wirusy, gdzie wysoki stopień zmienności może utrudniać zaprojektowanie ogólnego testu22.
Projektowanie sond
Podobnie jak w przypadku starterów PCR, projektowanie sond qPCR również zależy w dużej mierze od kontekstu sekwencji i pożądanego zastosowania. Pojedyncze sondy, takie jak Dual-Labeled Probes lub Molecular Beacons, mają zazwyczaj długość 20-30 zasad. Sondy Scorpions® mają krótsze sondy o długości 15-25 zasad. W systemie LightCycler lub FRET istnieją dwie sondy: sensor (sonda 1) i sonda kotwicząca (sonda 2), które znajdują się w bliskiej odległości, oddzielone 1-5 zasadami.
- Przy stosowaniu sond podwójnie znakowanych, Tm powinna być o 7 °C do 10 °C wyższa niż starterów, aby zapewnić, że sonda związała się z celem przed hybrydyzacją i wydłużeniem starterów. Dotyczy to również obu sond FRET w reakcji. W przypadku stosowania do wykrywania SNP, sonda czujnikowa (sonda 1) znajduje się nad miejscem niedopasowania, unikając końcowych 3 zasad sondy i ma niższy Tm (około 5 °C) niż sonda kotwicząca (sonda 2). Sonda Scorpions® stanowi wyjątek od tego zalecenia, ponieważ sonda wiąże się z nowo zsyntetyzowaną matrycą po wydłużeniu, a nie przed, jak w przypadku innych systemów sond.
- W przypadku badań ilościowych z użyciem podwójnie znakowanych sond należy dążyć do tego, aby sonda była umieszczona blisko 3' startera, ale nie zachodziła na siebie (około pięciu zasad); w celu wykrycia SNP należy umieścić podwójnie znakowaną sondę lub Molecular Beacon w środku amplikonu, a SNP w środku sondy.
- Unikaj guanidyny na końcu 5' sondy, obok reportera, ponieważ powoduje to wygaszanie.
- Upewnij się, że w sekwencji sondy jest mniej G niż C.
- Unikaj sekwencji o tej samej podstawie (<4) i sekwencji palindromicznych.
- Upewnij się, że sonda nie może przyjąć struktury drugorzędowej.
- Upewnij się, że sonda nie może hybrydyzować ze starterami.
- Przy projektowaniu sond do reakcji multipleksowych, upewnij się, że nie ma potencjalnych interakcji między sondami i starterami.
Sondy molekularne
.Po wybraniu odpowiedniego regionu sondy, do końców 5' i 3' dodawane są komplementarne trzony, aby utworzyć strukturę Molecular Beacon20. Poniższy przykład pokazuje dodanie sekwencji macierzystej (w kolorze czerwonym) do podwójnie znakowanej sondy w celu utworzenia Molecular Beacon (Rysunek 6.9 zaadaptowany z Thelwell 200021).

Rysunek 6.9.Adaptacja testu z podwójnie znakowaną sondą do formatu Molecular Beacon. Nucleic acids research by Oxford University Press. Powielono za zgodą Oxford University Press w formacie do ponownego wykorzystania w książce/e-booku za pośrednictwem Copyright Clearance Center.
Sondy Scorpions® Probes
.Sonda Scorpions® Probe wymaga połączenia sondy ze starterem do przodu w taki sposób, aby przyjęły one strukturę: 5' dyestem-probe-stem-quencher-blocker-primer. Starter i sonda muszą znajdować się na przeciwnych niciach, ponieważ sonda wiąże się z nowo utworzoną matrycą, która znajduje się na tej samej nici co starter. Przykład pokazany na Rysunku 6.10 przedstawia dodanie sekwencji znakującej, wygaszającej, blokującej PCR i macierzystej do podwójnie znakowanej sondy w celu utworzenia sondy Scorpions® (zaadaptowane z Thelwell 200021).

Rysunek 6.10.Adaptacja testu z podwójnie znakowaną sondą do formatu sondy Scorpions®. Badania nad kwasami nukleinowymi przez Oxford University Press. Powielono za zgodą Oxford University Press w formacie do ponownego wykorzystania w książce/e-booku za pośrednictwem Copyright Clearance Center.
Modyfikacje sond
Gdy sondy znajdują się w regionie sekwencji o niepożądanej heterogeniczności, niejednoznaczne zasady są zarządzane w sposób opisany dla starterów PCR. Podobnie, zablokowany kwas nukleinowy może być dodawany do sond z tych samych powodów, dla których jest dodawany do starterów.
Podwójnie znakowana sonda 5', Molecular Beacon lub Scorpions® Probe jest znakowana fluoroforem, zwykle 6-FAM™ dla pojedynczych testów lub w przypadku multipleksowania. dla pojedynczych testów lub podczas multipleksowania, zazwyczaj wybierając je w kolejności FAM, HEX™/JOE™, Cyanine 5 (kluczowe jest określenie kompatybilności etykiety z urządzeniem). Koniec 3' sondy podwójnie znakowanej lub Molecular Beacon oraz koniec 3' wewnętrznego regionu trzonu sondy Scorpions® Probe są modyfikowane cząsteczką wygaszacza. Historycznie, barwnik TAMRA był używany jako akceptor dla emisji FAM, co powodowało wygaszanie FAM. Rozwój technologii ciemnych wygaszaczy zaowocował powszechnym przyjęciem Black Hole Quenchers™ oraz niedawnym wprowadzeniem naszej kolekcji Onyx Quencher™ (patrz Quantitative PCR and Digital PCR Detection Methods).
Template Controls
Jedną z zalet stosowania stosunkowo krótkich amplikonów, które zazwyczaj mają mniej niż 150 zasad, jest możliwość syntezy długiego oligonukleotydu, który można wykorzystać jako syntetyczny cel amplifikacji. Zastosowanie takiego celu może pomóc w opracowaniu i optymalizacji testów, w których zamierzony cel może być rzadki lub może go brakować, na przykład w teście kontroli inhibicji SPUD23 (patrz Załącznik A) lub badania wykrywania chorób zakaźnych19.
Synteza i obsługa oligo./h2> Przy zamawianiu niestandardowych oligo do zastosowań PCR należy podjąć decyzje dotyczące pożądanej wydajności/skali syntezy, czystości i wymaganych modyfikacji. Każdy z tych czynników wpływa na inne, np, wyższy poziom oczyszczania spowoduje lepszą jakość oligonukleotydu, ale kosztem zmniejszenia ogólnej wydajności. Tabele 6.4, 6.5 i 6.6 zawierają wskazówki dotyczące skali syntezy i oczekiwanej wydajności produkowanych przez nas oligonukleotydów.
Oczyszczanie oligonukleotydów<Podczas syntezy DNA, każdy nukleotyd jest sekwencyjnie przyłączany do rosnącego łańcucha, zaczynając od 3' końca sekwencji. W każdym cyklu sprzęgania niewielki procent łańcuchów oligo nie zostanie przedłużony, co skutkuje mieszaniną produktu o pełnej długości i skróconych sekwencji. Po odłączeniu oligo od nośnika i usunięciu grup zabezpieczających, stosuje się oczyszczanie w celu oddzielenia produktu o pełnej długości od skróconych sekwencji. Ogólnie rzecz biorąc, czystość wymagana dla konkretnego zastosowania zależy od potencjalnego wpływu obecności skróconych oligomerów. W przypadku niektórych zastosowań kluczowe znaczenie ma obecność wyłącznie oligomerów pełnej długości (n). W przypadku innych, takich jak startery PCR, obecność krótszych oligomerów (n-1, n-2,...) może nie mieć wpływu na wyniki eksperymentu.
Oczyszczanie przez odsalanie
Procedura odsalania usuwa resztkowe produkty uboczne, które pozostają z etapów syntezy, rozszczepiania i deprotekcji.
Dla wielu zastosowań, w tym PCR, oczyszczanie przez odsalanie jest dopuszczalne dla oligos, które mają nie więcej niż 35 zasad, ponieważ przeważająca obfitość oligo o pełnej długości przeważa nad wszelkimi krótszymi produktami. Oligo wymagane do klonowania lub o długości większej niż 35 zasad wymagają dodatkowej metody oczyszczania, takiej jak oczyszczanie w odwróconej fazie (RP1), HPLC lub PAGE (w zależności od długości).
Oczyszczanie na kasecie z odwróconą fazą (RP1)
Separacja na kasecie z odwróconą fazą usuwa wysoki odsetek skróconych sekwencji. Różnica w hydrofobowości między produktem o pełnej długości a sekwencjami skróconymi jest wykorzystywana jako podstawa separacji. Podczas gdy oligo o pełnej długości jest zatrzymywane na kolumnie, skrócone sekwencje są wymywane. Pożądany produkt o pełnej długości jest następnie wymywany i usuwany z wkładu.
HPLC w odwróconym układzie faz
Wraz ze wzrostem długości oligo, udział sekwencji skróconych ma tendencję do zwiększania się. Nie wszystkie te zanieczyszczenia zostaną usunięte przez RP1, a zatem w przypadku dłuższych oligos, takich jak oligos szablonu sztucznego amplikonu lub oligos znakowanej sondy, zaleca się oczyszczanie HPLC lub PAGE. Wysokosprawna chromatografia cieczowa w odwróconej fazie (RP-HPLC) działa na tej samej zasadzie, co kaseta w odwróconej fazie. Jednak wyższa rozdzielczość pozwala na uzyskanie wyższych poziomów czystości. HPLC jest skuteczną metodą oczyszczania oligos z fluoroforami, takimi jak sondy qPCR, ponieważ ich wewnętrzna lipofilowość zapewnia doskonałe oddzielenie produktu od zanieczyszczeń. Ponadto, RP-HPLC jest metodą z wyboru dla większych skal ze względu na pojemność i właściwości rozdzielcze kolumny. Rozdzielczość oparta na lipofilowości zmniejsza się wraz ze wzrostem długości oligo. Dlatego RP-HPLC zwykle nie jest zalecana do oczyszczania produktów dłuższych niż 50 zasad. Chociaż dłuższe oligo (do 80 zasad) mogą być oczyszczane przy użyciu tej metody, może to mieć negatywny wpływ na czystość i wydajność.
HPLC z wymianą anionów
Rozdzielanie z wymianą anionów opiera się na liczbie grup fosforanowych w cząsteczce. Metoda oczyszczania anionowymiennego polega na zastosowaniu elucji z gradientem soli na czwartorzędowej amonowej kolumnie fazy stacjonarnej lub podobnej strukturze. Rozdzielczość jest doskonała do oczyszczania mniejszych ilości. Technika ta może być połączona z oczyszczaniem za pomocą RP-HPLC, dodając drugi wymiar do procesu separacji. HPLC z wymianą anionów jest ograniczona długością oligonukleotydu (zwykle do 40 merów). Im dłuższe oligonukleotydy, tym niższa rozdzielczość na kolumnie anionowymiennej HPLC, a tym samym niższa czystość docelowego oligo.
Elektroforeza w żelu poliakrylamidowym (PAGE)
Podstawą separacji PAGE jest ładunek nad masą cząsteczkową, co prowadzi do dobrej rozdzielczości wielkości, co skutkuje poziomem czystości 95-99% produktu o pełnej długości. Wydajność PAGE jest niższa niż w przypadku innych metod ze względu na złożone procedury wymagane do ekstrakcji oligos z żelu i usunięcia zdecydowanej większości skróconych produktów. Technika ta jest zalecana, gdy wymagany jest wysoce oczyszczony produkt. PAGE jest zalecaną metodą oczyszczania dla dłuższych oligos (≥50 zasad).
Podczas syntezy DNA, każdy nukleotyd jest sekwencyjnie przyłączany do rosnącego łańcucha, zaczynając od 3' końca sekwencji. W każdym cyklu sprzęgania niewielki procent łańcuchów oligo nie zostanie przedłużony, co skutkuje mieszaniną produktu o pełnej długości i skróconych sekwencji. Po odłączeniu oligo od nośnika i usunięciu grup zabezpieczających, stosuje się oczyszczanie w celu oddzielenia produktu o pełnej długości od skróconych sekwencji. Ogólnie rzecz biorąc, czystość wymagana dla konkretnego zastosowania zależy od potencjalnego wpływu obecności skróconych oligomerów. W przypadku niektórych zastosowań kluczowe znaczenie ma obecność wyłącznie oligomerów pełnej długości (n). W przypadku innych, takich jak startery PCR, obecność krótszych oligomerów (n-1, n-2,...) może nie mieć wpływu na wyniki eksperymentu.
Oczyszczanie przez odsalanie
Procedura odsalania usuwa resztkowe produkty uboczne, które pozostają z etapów syntezy, rozszczepiania i deprotekcji.
Dla wielu zastosowań, w tym PCR, oczyszczanie przez odsalanie jest dopuszczalne dla oligos, które mają nie więcej niż 35 zasad, ponieważ przeważająca obfitość oligo o pełnej długości przeważa nad wszelkimi krótszymi produktami. Oligo wymagane do klonowania lub o długości większej niż 35 zasad wymagają dodatkowej metody oczyszczania, takiej jak oczyszczanie w odwróconej fazie (RP1), HPLC lub PAGE (w zależności od długości).
Oczyszczanie na kasecie z odwróconą fazą (RP1)
Separacja na kasecie z odwróconą fazą usuwa wysoki odsetek skróconych sekwencji. Różnica w hydrofobowości między produktem o pełnej długości a sekwencjami skróconymi jest wykorzystywana jako podstawa separacji. Podczas gdy oligo o pełnej długości jest zatrzymywane na kolumnie, skrócone sekwencje są wymywane. Pożądany produkt o pełnej długości jest następnie wymywany i usuwany z wkładu.
HPLC w odwróconym układzie faz
Wraz ze wzrostem długości oligo, udział sekwencji skróconych ma tendencję do zwiększania się. Nie wszystkie te zanieczyszczenia zostaną usunięte przez RP1, a zatem w przypadku dłuższych oligos, takich jak oligos szablonu sztucznego amplikonu lub oligos znakowanej sondy, zaleca się oczyszczanie HPLC lub PAGE. Wysokosprawna chromatografia cieczowa w odwróconej fazie (RP-HPLC) działa na tej samej zasadzie, co kaseta w odwróconej fazie. Jednak wyższa rozdzielczość pozwala na uzyskanie wyższych poziomów czystości. HPLC jest skuteczną metodą oczyszczania oligos z fluoroforami, takimi jak sondy qPCR, ponieważ ich wewnętrzna lipofilowość zapewnia doskonałe oddzielenie produktu od zanieczyszczeń. Ponadto, RP-HPLC jest metodą z wyboru dla większych skal ze względu na pojemność i właściwości rozdzielcze kolumny. Rozdzielczość oparta na lipofilowości zmniejsza się wraz ze wzrostem długości oligo. Dlatego RP-HPLC zwykle nie jest zalecana do oczyszczania produktów dłuższych niż 50 zasad. Chociaż dłuższe oligo (do 80 zasad) mogą być oczyszczane przy użyciu tej metody, może to mieć negatywny wpływ na czystość i wydajność.
HPLC z wymianą anionów
Rozdzielanie z wymianą anionów opiera się na liczbie grup fosforanowych w cząsteczce. Metoda oczyszczania anionowymiennego polega na zastosowaniu elucji z gradientem soli na czwartorzędowej amonowej kolumnie fazy stacjonarnej lub podobnej strukturze. Rozdzielczość jest doskonała do oczyszczania mniejszych ilości. Technika ta może być połączona z oczyszczaniem za pomocą RP-HPLC, dodając drugi wymiar do procesu separacji. HPLC z wymianą anionów jest ograniczona długością oligonukleotydu (zwykle do 40 merów). Im dłuższe oligonukleotydy, tym niższa rozdzielczość na kolumnie anionowymiennej HPLC, a tym samym niższa czystość docelowego oligo.
Elektroforeza w żelu poliakrylamidowym (PAGE)
Podstawą separacji PAGE jest ładunek nad masą cząsteczkową, co prowadzi do dobrej rozdzielczości wielkości, co skutkuje poziomem czystości 95-99% produktu o pełnej długości. Wydajność PAGE jest niższa niż w przypadku innych metod ze względu na złożone procedury wymagane do ekstrakcji oligos z żelu i usunięcia zdecydowanej większości skróconych produktów. Technika ta jest zalecana, gdy wymagany jest wysoce oczyszczony produkt. PAGE jest zalecaną metodą oczyszczania dla dłuższych oligos (≥50 zasad).
*Gwarancja dotyczy oligosów 20-merowych lub dłuższych. Krótsze oligo mogą mieć mniej OD.
*Gwarancja dotyczy oligosów 20-merowych lub dłuższych. Krótsze oligo mogą mieć mniej OD.
Uwaga: Modyfikacje po syntezie mogą dać 50% mniej niż podane powyżej wartości.
Wszystkie sondy są oczyszczane metodą RP-HPLC.
Przygotowanie oligonukleotydów
Oligonukleotydy DNA dostarczone w stanie suchym są gotowe do użycia po ponownym zawieszeniu. Zaleca się ponowne zawieszenie oligonukleotydów w słabym buforze, takim jak TE (10 mM Tris, pH 7,5-8,0, 1 mM EDTA). W zastosowaniach, w których TE nie jest odpowiedni, można użyć sterylnej wody wolnej od nukleaz. Jednak woda wysokiej jakości może być lekko kwaśna i nie jest zalecana do długotrwałego przechowywania oligonukleotydów.
Roztwór podstawowy 100 μM można uzyskać, stosując następujące wytyczne: Weź liczbę dostarczonych nanomoli (nmol) (informacja znajduje się na etykiecie probówki i/lub dokumencie zapewnienia jakości dostarczonym wraz z oligo) i pomnóż przez 10. Wynik daje liczbę mikrolitrów płynu, którą należy dodać do probówki, aby uzyskać końcowe stężenie 100 μM. Na przykład, jeśli wydajność oligo wynosi 43,5 nmol, objętość do dodania dla 100 μM wynosi 435 μL. Należy zauważyć, że jest to odpowiednik roztworu podstawowego o stężeniu 100 pmol/μL. Roztwór podstawowy można następnie rozcieńczyć w razie potrzeby, w zależności od wymagań aplikacji. W przypadku PCR zwykle stosuje się stężenie robocze 10 μM lub 20 μM. Roztwór podstawowy należy przechowywać w podwielokrotnościach w temperaturze -20 °C i unikać wielokrotnych cykli zamrażania-rozmrażania.
Referencje
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?