Chromatograficzne oczyszczanie plazmidowego DNA (pDNA) do zastosowań w mRNA, szczepionkach DNA opartych na plazmidzie i wektorach wirusowych
- Role of Chromatography in Plasmid DNA Production
- Rodzaje chromatografii dostępne w produkcji plazmidowego DNA
- Wysokie stężenie soli maksymalizuje wychwyt pDNA
- Wyzwania związane z oczyszczaniem pDNA przy użyciu konwencjonalnych żywic chromatograficznych
- High-throughput Capture Using Anion Exchange Chromatography
- Case Study 1: Zwiększenie stężenia soli poprawia czystość pDNA podczas chromatografii anionowymiennej (AEX) poprzez usuwanie zanieczyszczających RNA
- Studium przypadku 2: Optymalizacja płukania anionowego
- Studium przypadku 3: Porównanie wydajności wymiany anionowej: Żywica vs. Membrana
Powszechne zastosowanie pDNA w szczepionkach i terapii genowej napędza zwiększony popyt, a w odpowiedzi produkcja plazmidów musi stać się bardziej wydajna i produktywna. Intensyfikacja etapów chromatograficznych może pomóc sprostać temu zapotrzebowaniu.
Rola chromatografii w produkcji plazmidowego DNA
Oczyszczanie pDNA jest trudne, ponieważ wiele krytycznych zanieczyszczeń ma ładunek ujemny, podobną wielkość i podobny poziom hydrofobowości. Końcowy masowy pDNA musi spełniać specyfikacje jakościowe określone przez agencje regulacyjne dotyczące czystości i musi zawierać ponad 90% pDNA w superzwiniętej izoformie. Etapy chromatografii są wykorzystywane do oczyszczania pDNA tak, aby spełniał te standardy regulacyjne. Jest ona stosowana zarówno jako etap procesu w celu osiągnięcia większej czystości, jak i jako narzędzie analityczne do monitorowania jakości pDNA.
Rodzaje chromatografii dostępne w produkcji plazmidowego DNA
Najczęściej stosowanymi technikami oczyszczania plazmidów są chromatografia jonowymienna (AEC) i chromatografia oddziaływań hydrofobowych (HIC). Obie techniki zostały wdrożone do wychwytywania lub pośredniego oczyszczania/polerowania i często są łączone.1,2 Chromatografia wykluczania (SEC) jest czasami włączana jako część schematu dalszych etapów; jest ona zwykle wybierana jako ostatni etap ze względu na jej wady w postaci niskiej przepustowości i powolnej kinetyki.1,3 HIC jest w stanie oddzielić natywne superzwinięte pDNA od izoform pDNA od bardziej hydrofobowych zanieczyszczeń kwasami nukleinowymi (RNA, genomowe DNA, zdenaturowane pDNA) i od endotoksyn.4 AEC osiąga usuwanie białek, RNA o niskiej masie cząsteczkowej i endotoksyn, ale jego skuteczność jest wysoce zależna od składu próbki.
Wysokie stężenie soli maksymalizuje wychwytywanie pDNA
W celu maksymalizacji wychwytywania pDNA należy zastosować wysokie stężenie soli w ładunku.1,5
Aby to zademonstrować, użyliśmy standardowego wsadu jako materiału wyjściowego do oczyszczania z oryginalnego E. coli lizatu, 3 sklarowanego przez odwirowanie i późniejszą filtrację wgłębną, a następnie bezpośrednio uzupełnionego NaCl.
Solenie sklarowanej paszy przed chromatografią wychwytu anionowymiennego jest szczególnie użytecznym podejściem do usuwania zanieczyszczeń RNA bez konieczności stosowania RNazy. Gatunki RNA słabiej wiążą się z nośnikami anionowymiennymi w porównaniu do silniej naładowanego pDNA. Dlatego uzupełnienie soli do optymalnej przewodności pozwoli zanieczyszczeniom RNA przepływać przez adsorbent AEX bez wiązania, podczas gdy pDNA wiąże się i jest następnie wymywane z wysoką czystością.
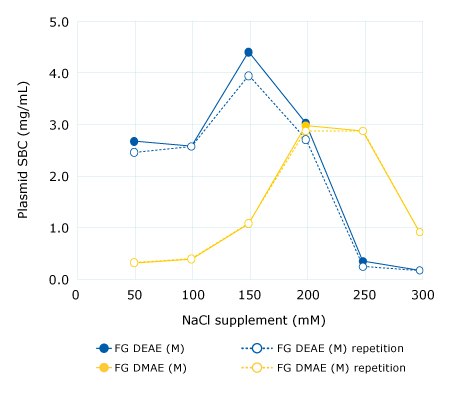
Rysunek 1.Test wsadowy w celu określenia optymalnego stężenia NaCl dla suplementacji lizatu. Statyczne zdolności wiązania (SBC) mierzono w 96-dołkowych płytkach filtracyjnych (1 ml na studzienkę). Wsad plazmidowy stanowił oryginalny oczyszczony lizat (pH 5,0, 67 mS/cm) uzupełniony rosnącymi stężeniami NaCl. FG = żywica Fractogel® EMD.
Optymalne stężenie soli do suplementacji musi zostać wstępnie określone przed rozpoczęciem oczyszczania dla każdej żywicy/adsorbera membranowego. Odbywa się to poprzez pomiar zdolności wiązania plazmidu przy rosnących stężeniach chlorku sodu. Zasada ta została zademonstrowana na przykładzie Fractogel® EMD DEAE (M) i żywic Fractogel® EMD DMAE (M) na Rysunku 1. W Studium przypadku 1 pokazujemy również, jak stężenie soli wpływa na wydajność, czystość i odzyskiwanie pDNA.
Wyzwania związane z oczyszczaniem pDNA za pomocą konwencjonalnych żywic chromatograficznych
W przypadku konwencjonalnych żywic chromatograficznych oczyszczanie pDNA stanowi wyzwanie, ponieważ jest ono znacznie większe niż białka, dla których żywice zostały pierwotnie zaprojektowane. Rozmiar pDNA uniemożliwia mu wejście w strukturę porów kulek żywicy, powodując niską zdolność wiązania i powolny transfer masy.4,5 Dodatkowo (większy) rozmiar zwiększa spadek ciśnienia i czas przetwarzania ze względu na lepkość paszy, niską rozdzielczość izoform i potencjalne zanieczyszczenie. Pomimo tych wad, większość istniejących procesów na dużą skalę wykorzystuje żywice chromatograficzne ze względu na ich stosunkowo wysoką rozdzielczość.
Producenci pDNA podejmują się jednak intensyfikacji etapów chromatografii przy użyciu mediów konwekcyjnych (tj. membran, monolitów i technologii opartych na włóknach) w celu poprawy wydajności i zwiększenia ogólnej wydajności.
Wysokoprzepustowe wychwytywanie przy użyciu chromatografii anionowymiennej
Ponieważ wysokoprzepustowe wychwytywanie przy użyciu chromatografii anionowymiennej jest szeroko stosowane w produkcji plazmidowego DNA, przeprowadziliśmy kilka badań optymalizacyjnych przy użyciu naszego membran chromatograficznych Natrix® Q.
Membrany chromatograficzne Natrix® Q zapewniają znaczną poprawę wydajności w porównaniu z tradycyjnymi żywicami. Wydajność (g/L/godz.) znacznie wzrasta, ponieważ kanały konwekcyjne utworzone przez makroporowatą strukturę polimeru mają wysoką gęstość miejsc wiązania czwartorzędowych amin z szybkim transferem masy. Te miejsca wiązania są dostępne nawet dla bardzo dużych cząsteczek docelowych, takich jak pDNA, przy dużych prędkościach przepływu, przy jednoczesnym zachowaniu celów oczyszczania wymaganych do produkcji GMP. Przy zdolności wiązania pierwotnego wychwytu wynoszącej ~10 mg/ml z oczyszczonego lizatu uzupełnionego NaCl, membrana chromatograficzna Natrix® Q może usunąć ponad 95% początkowego dużego RNA. W jednym z przykładów, membrana do chromatografii Natrix® Q osiągnęła ≥80% czystego plazmidowego DNA, z ~10% resztkowego RNA (na bazie A260) i wydajnością ~80% przy zaledwie 30 minutach czasu pracy.
Studium przypadku 1: Zwiększenie stężenia soli poprawia czystość pDNA podczas chromatografii anionowymiennej (AEX) poprzez usuwanie zanieczyszczających RNA
Cel
Optymalizacja stężenia NaCl w materiale ładunkowym w celu promowania wychwytywania produktu plazmidowego DNA, podczas gdy zanieczyszczenia RNA są usuwane w przepływie ładunku.
Materiały
- Urządzenie - Natrix® Q Micro 0.2 mL. Do każdego testu użyto nowego urządzenia Natrix® Q.
Warunki klarowania lizatu
- 6,5 kbp pDNA, miano 24 µg/ml. 1,5 M bufor K-octanowy, pH 5,3, 86,9 ms/cm
- Zawartość kwasu nukleinowego: 3,8% pDNA, 96,2% RNA. Zawartość endotoksyn: 380 000 EU/mg pDNA
- Oczyszczony lizat został załadowany do 11 mg pDNA/mL membrany przy trzech różnych przewodnościach obciążenia.
- Test 1: Nie dodano dodatkowej soli.
- Test 2: 35 mM NaCl zostało dodane do oczyszczonego lizatu
- Test 3: 75 mM NaCl zostało dodane do oczyszczonego lizatu
Analizy
- Zawartość DNA i RNA oceniana metodą HPLC (metoda Tosoh DNA-NPR)
- Zawartość endotoksyn oceniana testem Charles River Endosafe<
Wyniki
Wysoka zdolność wiązania na poziomie 11 mg plazmidu na ml membrany została osiągnięta, gdy do paszy nie dodano chlorku sodu. Jednak czystość plazmidowego DNA w eluacie była dość niska w tych warunkach - tylko 46% kwasu nukleinowego w eluacie stanowiło pDNA, a pozostałą część RNA. Wskazuje to, że wysoki procent RNA wiązał się z membraną w tych warunkach obciążenia.
Kiedy stężenie soli zostało zwiększone do 35 mM, nastąpiło nieznaczne zmniejszenie zdolności wiązania do 8 mg plazmidu na ml membrany. Jednak większość zanieczyszczeń RNA przepłynęła przez membranę przy tej podwyższonej przewodności, co skutkowało poprawą czystości (77% eluowanego kwasu nukleinowego stanowiło pDNA). Wysoka wydajność została również zaobserwowana w puli elucyjnej na poziomie 88%. Klasyfikacja endotoksyny została również osiągnięta dzięki tej strategii; pula elucyjna ma 3100 jednostek endotoksyny na mg plazmidu.
Kiedy stężenie NaCl zostało zwiększone do 75 mM, zaobserwowano znaczny spadek zdolności wiązania. Przy tym podwyższonym stężeniu ani RNA, ani plazmidowy produkt DNA nie są w stanie wystarczająco związać się z błoną anionowymienną (Rysunek 2). W związku z tym ustalono, że suplementacja lizatu 35 mM NaCl zapewnia najlepszą równowagę między wydajnością, czystością i odzyskiem na membranie Natrix® Q.
Suplementacja solą klarowanej paszy przed chromatografią wychwytu anionowymiennego jest szczególnie przydatnym podejściem do usuwania zanieczyszczeń RNA bez konieczności stosowania RNazy. Gatunki RNA słabiej wiążą się z nośnikami anionowymiennymi w porównaniu do silniej naładowanego pDNA. Dlatego uzupełnienie soli do optymalnej przewodności pozwoli zanieczyszczeniom RNA przepływać przez adsorbent AEX bez wiązania, podczas gdy pDNA wiąże się i jest następnie wymywane z wysoką czystością.
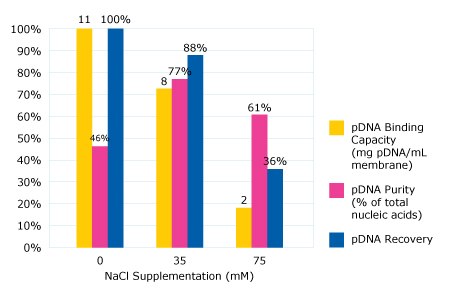
Figure 2.Impact of salt supplementation on capacity, purity, recovery of pDNA.
Studium przypadku 2: Optymalizacja płukania anionowymiennego
Cel
Ocena zdolności detergentu i buforów płuczących EDTA do dalszej poprawy Natrix® Q RNA i usuwania endotoksyn.
Materiały
- Urządzenie - Natrix® Q Micro 0,2 ml. Do każdego testu użyto nowego urządzenia Natrix® Q.
- Pasza - Oczyszczony i sterylnie przefiltrowany lizat pDNA, uzupełniony 35 mM NaCl
Aby zoptymalizować strategię płukania dla etapu wychwytywania anionowymiennego, kontrolne warunki płukania porównano z eksperymentalną strategią płukania detergentowego, w której do płukania dodano niskie stężenie detergentu, a także 2 mM EDTA do buforu do płukania i elucji. Podejście to poprawiło czystość kwasu nukleinowego z 77% do 95% i zmniejszyło stężenie endotoksyny do 500 jednostek endotoksyny na miligram plazmidu.
Studium przypadku 3: Porównanie wydajności wymiany anionowej: Żywica vs. Membrana
Cel
Porównanie obserwowanej wydajności chromatografii membranowej Natrix® z historycznymi danymi referencyjnymi dla żywicy chromatograficznej.
Materiały
- Adsorbery - Wydajność membrany do chromatografii Natrix® Q w porównaniu z historycznymi danymi referencyjnymi dla Fractogel® DMAE (patrz Tabela 6 dla danych referencyjnych chromatografii żywicznej)
- Podstawa zasilania - Rozważmy partię 300 l oczyszczonego lizatu zawierającego 7.1 g pDNA
Wyniki
Użycie membrany chromatograficznej Natrix® Q zwiększyło wydajność etapu chromatografii 24-krotnie, z 0,30 g pDNA/L/godz. do 7,3 g pDNA/L/godz. Zyski te można przypisać dwóm czynnikom:
- Pojemność wiązania pDNA dla membrany wynosi 8 g/L w porównaniu do zaledwie 3 g/L dla tradycyjnej żywicy; oraz
- Chromatografia membranowa oferuje bardzo szybkie szybkości przepływu ze względu na otwartą strukturę porów, umożliwiając czas cyklu nieco ponad 1 godzinę, podczas gdy wolniejszy proces żywicy może wymagać prawie 10 godzin na cykl. Można to wykorzystać do przeprowadzenia kilku cykli z chromatografią membranową w podejściu rapid cycling.
Dowiedz się więcej o procesie produkcji pDNA podczas naszego webinarium "Efektywne i wydajne projektowanie procesu oczyszczania plazmidowego DNA."
Wybór produktów i metod chromatograficznych może być wyzwaniem, ponieważ są one wysoce specyficzne dla procesu i często odgrywają kluczową rolę na wielu etapach, od wczesnego do późnego etapu. Aby uprościć to wyzwanie dla producentów biofarmaceutyków, dowiedz się więcej o naszym Portfolio chromatograficzne Emprove®, stworzone w celu zaspokojenia konkretnych potrzeb w zakresie usprawnionego wyboru i kwalifikacji żywicy.
Produkty
Referencje
Zaloguj się lub utwórz konto, aby kontynuować.
Nie masz konta użytkownika?